Successful case
科研解决方案

使用在目标性状上显著不同的双亲本构建性状分离群体,例如自交群体、回交群体、重组自交系群体等。
通过对双亲本群体进行基因型检测,结合表型数据对目标性状进行QTL定位。
QTL初定位一般基于低代的群体,能够得到一个比较大的定位区段。
QTL精细定位通过提高群体代数、缩小连锁区段、加密分子标记来逐渐缩小QTL定位区段,并最终完成基因定位与克隆。
与其他类型的基因定位方法例如GWAS等相比,双亲群体QTL定位有以下特点。
基于自然群体的GWAS分析需要面临由于群体结构而带来的定位结果假阳性问题,而双亲本QTL定位则不需要考虑群体结构问题,遗传背景噪音相对较小。
双亲本群体的构建相对直接,只需要找到在目标性状上有显著差异而且遗传背景最好不要太远的一对亲本即可,不需要进行大规模的自然群体收集。
QTL初定位由于分离区段相对较大不需要密度太高的分子标记,而QTL精细定位只需要在特定区段进行标记加密即可,所以双亲群体QTL定位所需要的分子标记检测量相对比较少。

1、提高双亲群体代数
2、在目标区段设计双亲多态性KASP标记
3、反复缩小定位区段直至找到功能基因

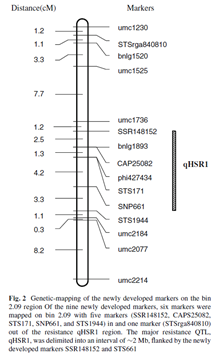
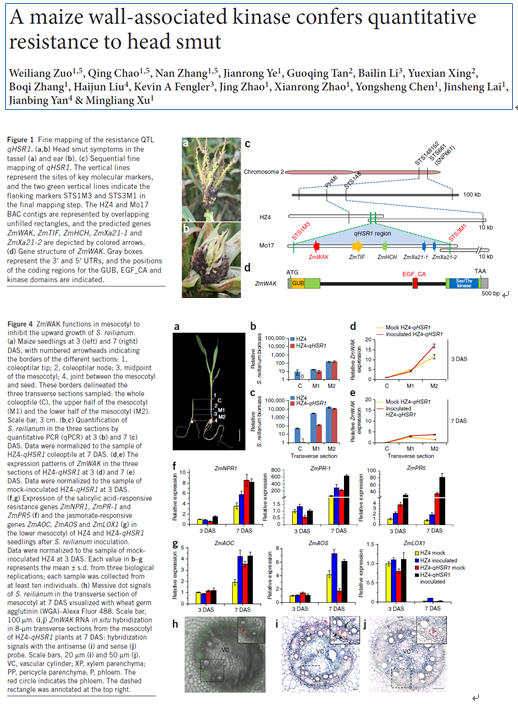
中国农业大学 徐明良 老师实验室基于一个双亲群体,对抗丝黑穗病基因 qHSR1 完成了从初定位、精细定位、品种改良、基因克隆的完整工作,最终的基因克隆文章发表于Nature Genetics期刊。
qHSR1 定位群体的双亲本是抗病亲本 Ji1037 和感病亲本黄早四。
文章Chen et al. 2008基于 BC2:3 和 BC2F2 群体将 qHSR1 初定位到 bin2.09 上大约 2Mb 的区段,后续工作进一步将 qHSR1 精细定位到了大约 152kb 的区段内。
另一篇文章Zhao et al. 2012描述了将 qHSR1 导入10个国内玉米自交系改良丝黑穗病抗性,结果显示在自交系和杂交种的层面上都带来了明显的抗性提升,与此同时大部分农艺性状没有发现影响。Zuo et al. 2014最终发表了 qHSR1 基因克隆与基因功能机制研究结果,该文发表在Nature Genetics期刊。