Successful case
科研解决方案

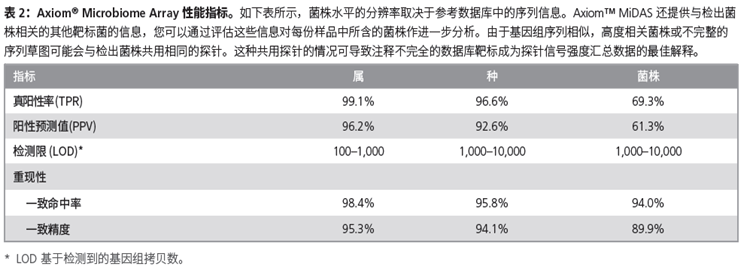
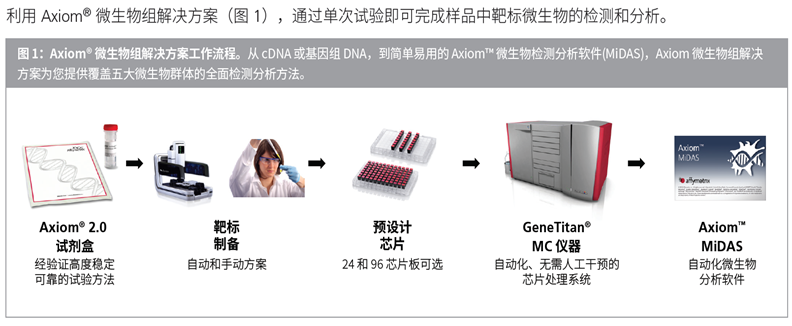
微生物组分析芯片(Microbiome Array),由 Affymetrix 与劳伦斯利福摩尔国家实验室(Lawrence Livermore National Laboratory)合作开发,利用简单易用的软件在可扩展的平台上实现菌种和菌株水平的检测。
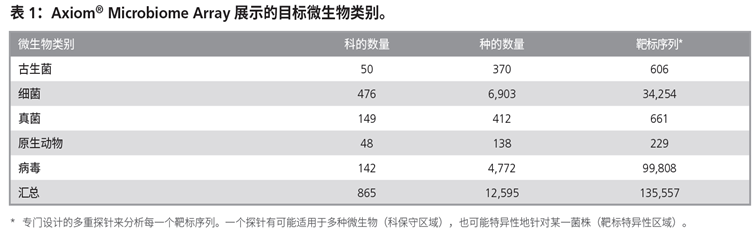
利用 Axiom Assay 生物化学技术,基于NCBI 数据库序列数据分析科保守区域和靶标特异性区域的非多态性序列,可检出 11,000 多种微生物,包括古生菌、细菌、真菌、原生动物和病毒,在单次试验即可检出样品中的所有已知微生物。
Axiom® Microbiome Array芯片内容对样品类型没有限制,适用于营养基因组学、农业基因组学和动物研究与建模。

1.芯片中含有针对 11,000多种微生物而设计的探针;
2.全面覆盖五大微生物群体:古生菌、细菌、真菌、原生动物和病毒;
3.可实现菌种和菌株水平的检测; RNA 病毒检测使用cDNA作为模板;
4.可扩展的平台,配合简单易用的分析软件;
5.芯片板有 24 和 96 两种规格可以选择;

1.营养基因组学研究(包括微生物群落对营养和饮食健康的影响)
A.探索人类肠道微生物群与疾病状态(例如,溃疡性结肠炎、肠道炎症和心血管疾病)之间的关联
B.对益生菌和益生元混合物进行高分辨率分析,来分析它们对人类肠道菌群的影响
2.农业基因组学
A.通过评估动物肠道微生物群落优化投喂
B.评估微生物群落对土壤生产力的影响
C.家畜健康状态检查
3.动物研究与建模
A.通过微生物分析检测病原体
B.动物园筛查(环境监测和动物菌落健康状态监测)
C.疾病预防
D.微生物状态和疗效评估
芯片有 24 和 96 两种制式可选,中玉金标记从收到样本到完成客户所需的分析需要一个月左右,如果客户需要定制化服务则可能需要更长的时间。
1. Khalil N. A., et al. In vitro batch cultures of gut microbiota from healthy and ulcerative colitis (UC) subjects suggest that sulphate-reducing bacteria levels are raised in UC and by a protein-rich diet. International Journal of Food Sciences and Nutrition 65(1):79–88 (2014).
2. Rath H. C., et al. Normal luminal bacteria, especially Bacteroides species, mediate chronic colitis, gastritis, and arthritis in HLA-B27/human ß2 microglobulin transgenic rats. The Journal of Clinical Investigation 98:945–953 (1996).
3. Tuohy K. M., et al. ‘The way to a man’s heart is through his gut microbiota’–dietary pro- and prebiotics for the management of cardiovascular risk. The Proceedings of the Nutrition Society 73(2):172–185 (2014).
4. Jonkers D., et al. Probiotics in the management of inflammatory bowel disease: a systematic review of intervention studies in adult patients. Drugs 72(6):803–823 (2013).
5. Jackson K. G., Lovegrove, J. A. Impact of probiotics, prebiotics and synbiotics on lipid metabolism in humans. Nutrition and Aging 1(3,4):181–200 (2012).
6. Myer P. R., et al. Gut bacterial communities and their association with production parameters in beef cattle. Journal of Animal Science 94(Supp 2):Abstract 392 (2016).
7. de Luna R. G., et al. Evaluation of pasture soil productivity in the semi-arid zone of Brazil by microbial analyses. Brazilian Journal of Microbiology 39(3):409–413 (2008).
8. Shanks, O. C., et al. Community structures of fecal bacteria in cattle from different animal feeding operations. Applied and Environmental Microbiology 77(9):2992–3001 (2011).
9. Mansfield K. G., et al. Workshop summary: detection, Impact, and control of specific pathogens in animal resource facilities. ILAR Journal 51(2):171–179 (2010).
10. Gardner S. N., et al. A microbial detection array (MDA) for viral and bacterial detection. BMC Genomics 11:668 (2010).
11. McLoughlin K. S. Microarrays for pathogen detection and analysis. Briefings in Functional Genomics 10(6):342–353 (2011).
